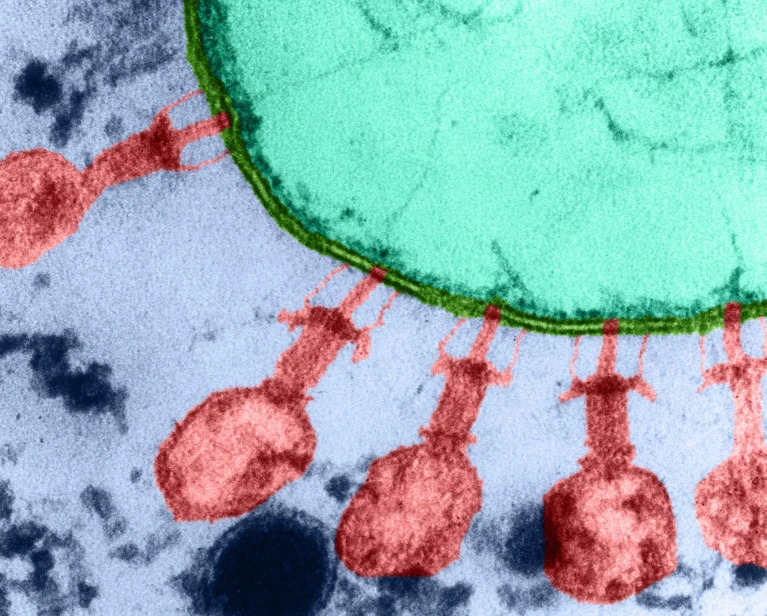
Американські науковці зі Стенфордського університету у Каліфорнії використали ШІ для створення вірусів, здатних відстежувати та вбивати штами кишкової палички Escherichia coli (E. coli).
“Це перший випадок, коли системи ШІ навчились створювати когерентні послідовності в масштабі геному. Наступний крок — це створення життя за допомогою ШІ. Ми сподіваємося, що подібна стратегія зможе доповнити наявні методи фагової терапії та в майбутньому розширити можливості терапії для боротьби з патогенами, що викликають занепокоєння“, — розповідає спеціаліст з обчислювальної біології зі Стенфорду Браян Хай.
До цього моделі ШІ вже використовувались для генерації послідовностей ДНК, окремих білків та багатокомпонентних комплексів. Однак проєктування повноцінного геному — набагато складніша задача, оскільки взаємодії між генами, а також реплікація та регуляція дуже складні.
Наразі ШІ здатен допомагати вченим маніпулювати найскладнішими біологічними системами, включно із цілими геномами. Для проєктування вірусних геномів дослідники використовували Evo 1 та Evo 2 — моделі штучного інтелекту, що аналізують та генерують послідовності ДНК, РНК та білків. Початково дослідникам був необхідний шаблон проєктування — послідовність, яка спрямовує модель штучного інтелекту до створення геному з бажаними характеристиками.
Було обрано бактеріофаг phi X 174 — одноланцюжковий ДНК-вірус, який містить 5 тис. 386 нуклеотидів в 11 генах та всі генетичні елементи для зараження господарів та реплікації всередині них. Моделі Evo 1 та Evo 2 були попередньо навчені на понад 2 млн фагових геномів. Окрім цього дослідники додатково навчили ШІ з використанням методу контрольованого навчання для створення вірусних геномів, подібних phi X 174, зі специфічною функцією зараження штамів E. coli, особливо стійких до антибіотиків.
Дослідники проаналізували тисячі послідовностей, згенерованих ШІ, та скоротили пошук до 302 життєздатних бактеріофагів. Більшість потенційних кандидатів мали понад 40% ідентичних з phi X 174 нуклеотидів, однак деякі з них мали зовсім інші кодувальні послідовності.
Дослідники синтезували ДНК з геномів, згенерованих моделями Evo 1 та Evo 2, та інтегрували цю ДНК в організм бактерій-господарів для вирощування фагів. Після цього фаги випробували на здатність інфікувати та знищувати E. coli.
За результатами випробувань, близько 16 з 302 бактеріофагів продемонстрували специфічність до E. coli і змогли інфікувати ці бактерії. Дослідники також виявили, що комбінації фагів, створених за допомогою ШІ, здатні інфікувати та знищувати 3 різні штами E. coli, на що був нездатний phi X 174.
Як зазначає спеціаліст з обчислювальної біології з лабораторії Колд-Спрінг-Харбор в Лорел-Холлоу, у штаті Нью-Йорк, Пітер Ку, однієї моделі Evo поки недостатньо для розробки та створення вірусів без сторонньої допомоги науковців. У науковій статті автори розглянули питання біологічної безпеки. Вони стверджують, що виключили віруси, які вражають еукаріотів, включаючи людину, з даних для навчання моделей Evo. Вивчені ними системи фагів phi X 174 та E. coli також не були патогенними і мають довгу історію безпечного використання в молекулярно-біологічних дослідженнях. Дослідники сподіваються, що їх підхід можна буде використовувати для безпечного створення розроблених за допомогою ШІ вірусів, які лікуватимуть різні захворювання та розв’язуватимуть проблеми громадської охорони здоров’я, включаючи проблему резистентності бактерій.
Результати дослідження опубліковані на сервері препринтів bioRxiv
Джерело: Nature










Повідомити про помилку
Текст, який буде надіслано нашим редакторам: